
No ano passado, pesquisadores encontraram mecanismos pelo qual o SARS-CoV-2, vírus causador da COVID-19, consegue escapar da nossa resposta imunológica. Esses mecanismos incluem a apresentação de um peptídeo mutante, que não ativa os linfócitos, e a produção de moléculas semelhantes ao MHC, que servem como isca para a ligação dos linfócitos e permitem a ação de espécies reativas de oxigênio na célula ligante.
Linfopenia é marcador de mau prognóstico na COVID-19
A pandemia causada pelo SARS-CoV-2 teve índices altos de mortalidade, que podem estar relacionados à linfopenia (redução da quantidade de linfócitos, a principal célula de defesa do nosso sistema imunológico) e à alteração na apresentação de antígenos pelo MHC.
A linfopenia dos pacientes com COVID-19 inclui redução das células T CD4+, células T CD8+, células B e células natural killer (NK), sendo o dano às células T CD8+ mais significativo.
A linfopenia no aparecimento inicial da COVID-19 está associada a um prognóstico ruim, sendo que sua presença e sua gravidade são preditores confiáveis do desfecho clínico da doença (incluindo mortalidade, necessidades de terapia intensiva e requisitos de oxigênio).

Vamos relembrar:
Para que os linfócitos possam atacar os microrganismos, eles precisam interagir com eles e reconhecê-los. Entretanto, os linfócitos T não conseguem interagir diretamente com os antígenos, precisando que estes sejam apresentados a eles. É aí que entram as moléculas do MHC, ou Complexo Principal de Compatibilidade. As moléculas de MHC se encontram na superfície das células e apresentam para os linfócitos o conteúdo daquela célula: caso a célula tenha sido infectada por um vírus, peptídeos desse vírus serão mostrados no MHC.
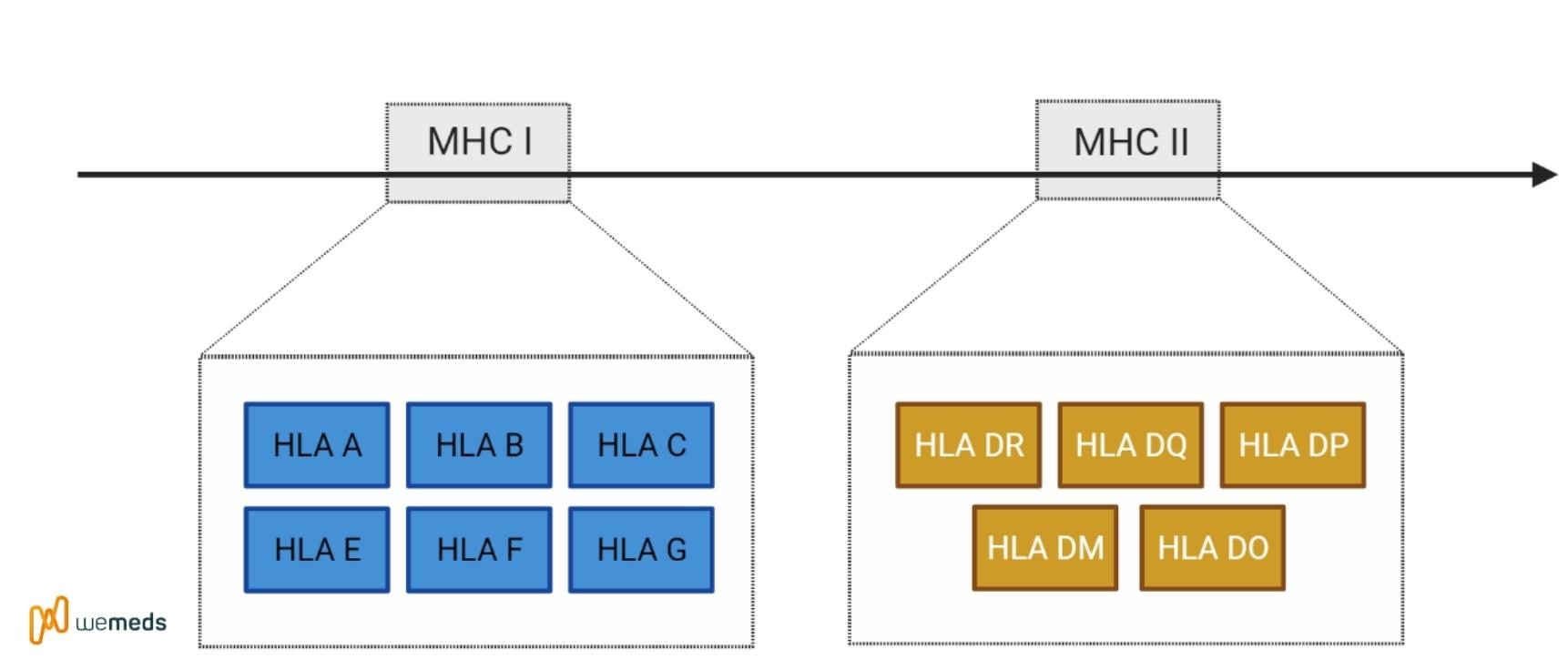
As moléculas de MHC podem ser de duas classes: MHC classe I e MHC classe II.
Além da linfopenia, a alta taxa de letalidade da COVID-19 está relacionada ao mau desempenho do MHC II.
A apresentação de MHC II para as células T é um pré-requisito essencial para a produção de anticorpos dependente de células T. O MHC de classe I deve apresentar os epítopos do SARS-CoV-2 com eficiência, pois isso afeta a imunidade das células T à infecção.

Como o SARS-CoV-2 escapa do nosso sistema imunológico?
Uma das maneiras pelas quais o SARS-CoV-2 evita a vigilância de células T CD8+ é através da mutação do epítopo que será apresentado pelo MHC I. Peptídeos mutantes exibem ligação reduzida ou ausente ao MHC I, o que faz com que os LT CD8 não reconheçam esse antígeno e não exerçam sua resposta funcional, fenômeno observado nas células T CD8+ isoladas de pacientes com COVID-19.
Além disso, o SARS-CoV-2 evoluiu uma estrutura semelhante ao MHC que pode se ligar ao receptor de MHC das células imunes. Os pesquisadores observaram que células imunes que não conseguiam se ligar à forma semelhante ao MHC do vírus sobreviveram.

Num estudo de pesquisadores chineses, foi utilizado o método de busca de domínio para descobrir que muitas proteínas do vírus SARS-CoV-2 têm estruturas semelhantes ao MHC. O vírus emprega as estruturas semelhantes ao MHC (da proteína S) como isca. Depois que a proteína SARS-CoV-2 S se liga às células CD4+ T, CD8+ T e NK, os radicais livres de oxigênio (ROS) gerados pela proteína E destroem essas células imunes, resultando em uma diminuição no número de linfócitos.
Ou seja, quando as células do sistema imune se ligam às células infectadas para reconhecer o antígeno, são atacadas pelas espécies reativas de oxigênio e morrem.
Papel das variantes genéticas
Mutações na região genética que codifica as estruturas semelhantes ao MHC das proteínas virais (como a proteína S) promoveram uma redução da resistência imune e uma transmissão mais robusta. Por exemplo:
- Mutações nas regiões 127-194 da proteína S foram a principal razão para a transmissão robusta das variantes delta;
- Mutações nas regiões 144-162 da proteína S regulavam a formação do trímero S;
- As mutações de RdRP: G671S e N: D63G da variante delta causaram carga viral alta;
- Mutações nas regiões 62-80 da proteína S das variantes alfa, beta e lambda foram um fator importante para a disseminação rápida;
- Mutações nas regiões 616-676 e 1014-1114 da proteína S foram causas de alta mortalidade para infecções por variantes gamma.


Conclusão
Os altos índices de mortalidade da COVID-19 estão relacionados aos mecanismos de escape do SARS-CoV-2 do sistema imunológico. Esses mecanismos incluem a apresentação de um peptídeo mutante, que não ativa os linfócitos, e a produção de moléculas semelhantes ao MHC, que servem como isca para a ligação dos linfócitos e permitem a ação de espécies reativas de oxigênio na célula ligante.
—
Referências:
LIU, W.; HUALAN, L. COVID-19: Attacks Immune Cells and Interferences With Antigen Presentation Through MHC-Like Decoy System. J Immunother 2023;46:75–88.